進化距離とは
「進化距離(evolutionary distance)」をご存知でしょうか。
「距離」という言葉は、広く使われている用法としては「家から病院までの距離」のように、その道の長さなどを測るために使われます。
が、広義では、「ある2つのモノはどのくらい違っているのか?」という数字の尺度と考えます。
「進化距離」とは、端的に言うと「ある2つの生物が、どれほど違う生物なのか」を数値で表現したものです。
「どれほど違う生物なのか」というのはつまり、古来の共通祖先から「どれほど進化してきたのか」と見なすことが出来るので、「進化距離」と呼ばれます。
これは主に分子系統学や生物進化学の分野で使用されます。
具体的には、「ヒトとサルはどれほど違うのか」を測りたい時には、それを「ヒトとサルは猿人からそれぞれどのくらい進化してきたか」と考え、
「猿人とヒトの違い」+「猿人とサルの違い」=「ヒトとサルの違い」と解釈します。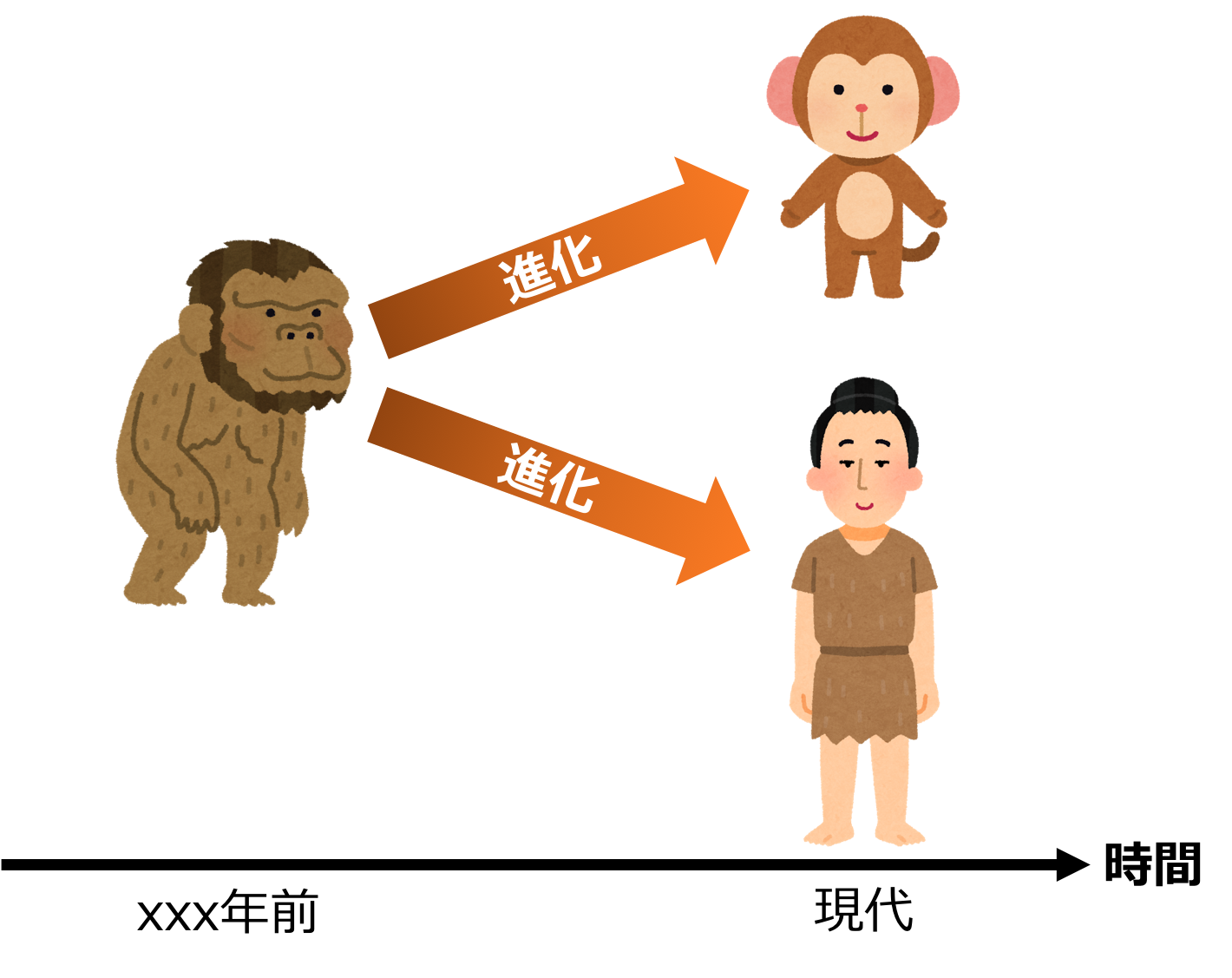
そして、進化距離は一般的にそれぞれの生物が持つDNAの情報を用いて計算されます。
(このあたりの更に細かい話は系統解析の記事をご参照ください。)

この記事では、DNA情報を「距離」の情報にするための具体的な計算方法を見ていきます。
DNA配列とは
まずは、DNA(デオキシリボ核酸)についてです。
生物学的な細かい説明は行いませんが、DNAとは地球上ほとんどの生物が持っている、生物の「遺伝情報」を保持する物質です。
これは「A(アデニン)」「T(チミン)」「G(グアニン)」「C(シトシン)」の4種類の物質(「塩基」と呼びます)が、ACGATCGTATCTGGATCGA….と何億個も連なって出来ています。
DNAが全く同じ生物というのはあり得ません。逆に、全く異なる生物でも、DNAの大半は同じという事も良くあります。マウスは99%がヒトと同じ、という報告もあります。
さて、なぜ「2つの生物のDNAが異なる」のかと言えば、「進化の過程でDNAが変異していっている」からだと考えられます。
共通祖先では同じ生物(同じDNA)だったのに、生物が分岐してからそれぞれでDNAが少しずつ変わっていき、今があると。
共通祖先のDNAについては知る由もありませんが、現存している2つの生物に関しては、それらのDNAの並びを見比べれば「2生物がどれほど異なるのか」ということは分かりそうです。
そして、「2生物がどれほど異なるのか」というのは、紛れもなく「進化距離」の定義です。
DNA配列の長さを揃える
では、ここで生物A・生物B・生物Cに登場してもらいます。それぞれのDNA配列が以下のようだったとしましょう。
(DNA配列は本来、何億個も連なっているものですが、便宜上短いものと仮定します。)
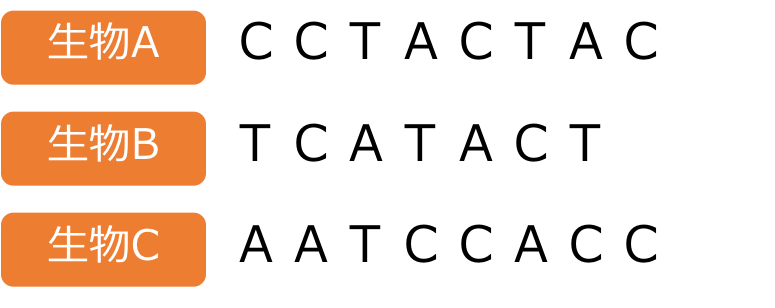
どうも、長さがまちまちのようです。このままだと、何が何と近いのか、遠いのか、比べづらいですね。
そんな時は「アライメント」という手法の出番です。
アライメントというのは、調べたいDNAの長さを揃える処理のことです。(DNAデータ以外にも用いられます。)
以下のように「ギャップ」と呼ばれる記号を適切な場所に埋め、なるべく全体の違いが少なくなるように調整します。

このように長さが揃っていると、どの生物とどの生物が近いのか、比べやすくなったかと思います。
アライメントの行い方にも沢山あるのですが、ここでは「あるアライメント手法を適用したら、上記のようになった」と考えてください。
では、ここから、具体的に「ある生物とある生物の近さ」を数値化していきましょう。
2つのDNA配列の距離を測る
さて、いよいよ進化距離の計算です。
進化距離の計算方法はたくさんあり、すべてを列挙することはできません。
今回は、最もシンプルな「P-distance(P距離)」と呼ばれる方法についてご紹介します。
ギャップの取扱い
進化距離を計算する前に、先程アライメントにおいて入った「ギャップ」の存在が気になります。
これはDNAの一部ではなく、人工的に仮定したものです。それをA,T,G,Cと同様に考えるのも変な気がします。
話が少し戻りますが、「2つの生物のDNAが異なる」ということは即ち、「進化の過程でDNAが変異していっている」からだと述べました。
ということは、「2つの生物のDNAの長さが違う」ということは即ち、進化の過程でDNAに挿入や欠失が発生しているということを表していると解釈できます。
ギャップの存在は、進化をより詳しく説明しているとも捉えられます。
・・・と、ギャップを含む・含まないにはいずれも一長一短があり、一概にどちらが良いとは言えません。
しかし、どうも「ギャップの入っている箇所は無いものとする」という計算が一般的に思われます。
(無論、一般的であるというだけで、それが正しいかどうかは別問題であるとはご理解下さい。)
ギャップの取り除き方にも2種類あり、「完全削除(complete deletion)」と「ペアワイズ削除(pairwise deletion)」があります。
完全削除は、比較したいすべての配列をタテに並べ、ひとつでもギャップがあったら、そのタテの列をすべて無しとする方法。
ペアワイズ削除は、比較する2つの配列をタテに並べ、ひとつでもギャップがあったら、その2つを比較する時のみにおいて、そのタテの列をすべて無しとする方法。
図にすると以下になります。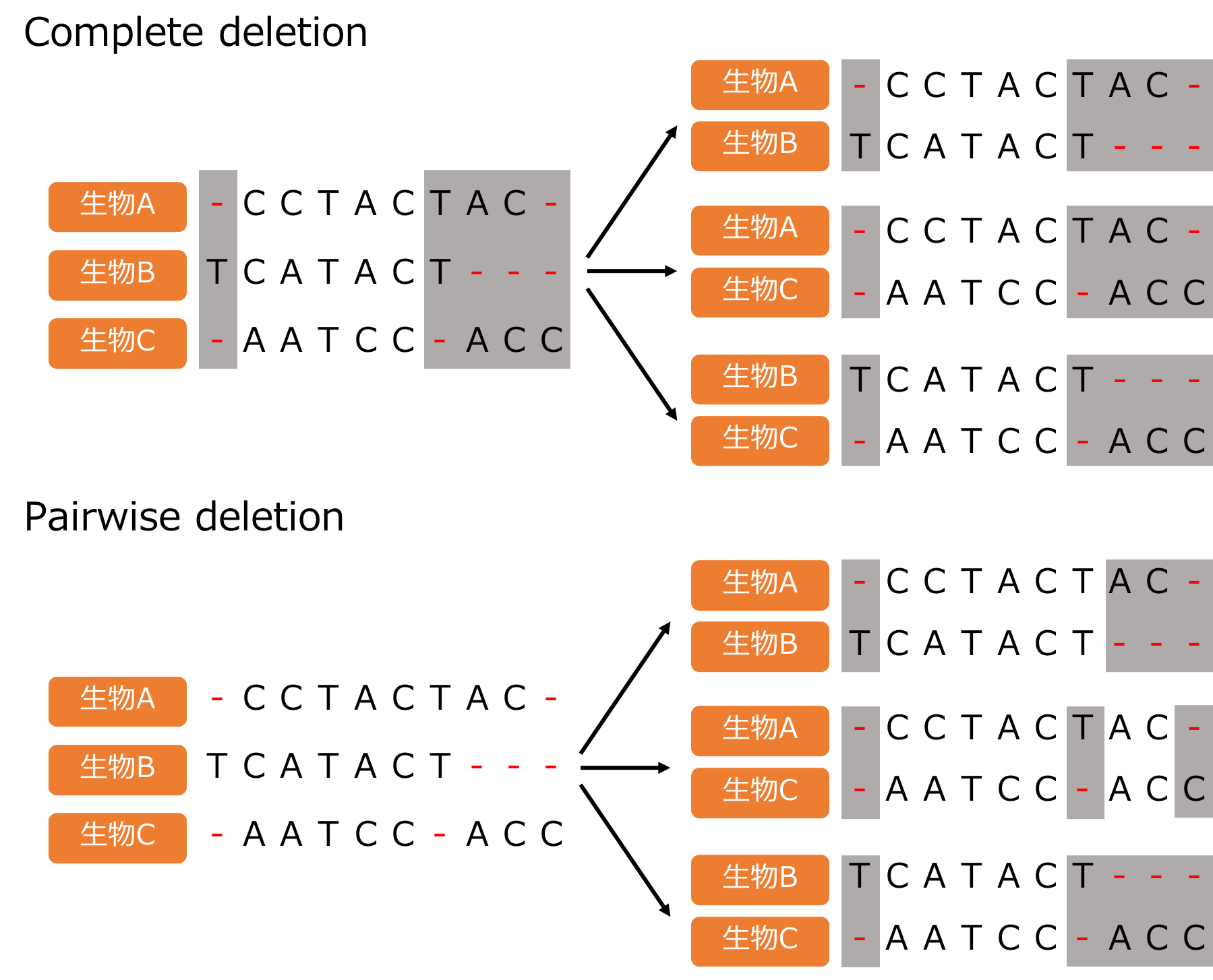
これについても、どちらが正しいとは言い切れません。
私としては、「生物AとBの比較」と「生物AとCの比較」のときで生物AのDNAの長さが違うという事に違和感を覚えるので「完全削除」を選ぶ場合が多いです。
が、「完全削除」にするとDNAの情報を多くカットしてしまうので、あまりにもそれによる情報量の欠落が大きい(ギャップの数が多い)場合は「ペアワイズ削除」にする事もあります。
そもそも、「ギャップを考慮して計算する」という手段を取った場合は完全削除かペアワイズ削除かで悩むことはないのですが・・・。
と、色々ありましたが、ここまでの事を決めれば後は計算するだけです。
今回は、ギャップは完全削除で扱っていきます。
P-Distance(P距離)で進化距離を求める
進化距離を計算する方法の中でも、P-distanceが最もシンプルな方法です。
これは、比べたい2つの配列をタテに見比べて、「異なっている箇所は全体の何%か?」という計算です。
完全に一致していたら0で、まったくバラバラならば1となり、数値が小さければ小さいほど、2つのDNAは近いと判断できます。
完全削除した配列を再掲します。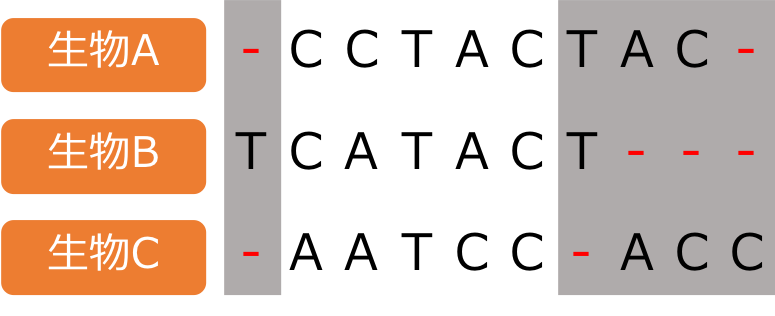
生物Aと生物Bは5文字中、1文字が異なります。
これはつまり、「祖先から生物Aへの変異」+「祖先から生物Bへの変異」の回数が1回であると推測している事になります。
P-distanceとしては、1/5=0.2です。
同様の計算で、
生物Aと生物Bの進化距離は0.2、
生物Aと生物Cの進化距離は0.6、
生物Bと生物Cの進化距離は0.4
・・・と計算できました。
このような計算を行うことにより、「ある生物とある生物の近さ」が数値化、つまり「進化距離」の計算ができました。
例えばこの数値を元にクラスタリングを行うと、進化距離が小さいほど「最近分岐した」と考えられるので、進化系統樹を作ることが出来ます。
今回示した方法は、おそらく最もシンプルと思われる方法で、おそらくは1960年くらいから確立されていた手法です。
今では更に細かい状況を加味した方法は山ほど出ています。
とはいえ、現代でも作業の流れとしては上記をおおよそ踏襲しております。
「進化距離」とは何なのか?どうやって計算するのか?・・・といった事をお伝えできていれば幸いです。
また、進化距離については、以下の書籍にも1章を割いて丁寧に解説されています。
専門的な内容となりますが、現在販売している日本語の書籍の中では最も充実しているのではないかと考えています。
※後日追記:「あるDNAが複数回置換している可能性」を考慮した「ポアソン距離」についての記事を作りました。